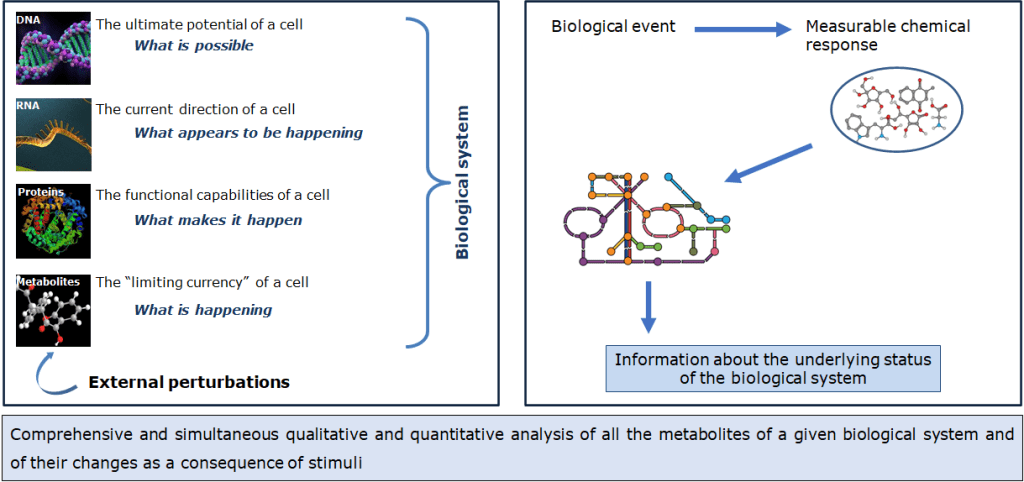
Nel contesto della metabolomica, gli approcci di analisi statistica più comuni si suddividono in metodi univariati e multivariati. Ogni metodo offre approfondimenti unici sulla struttura dei dati. L’analisi multivariata opera su una matrice di variabili e mette in evidenza le caratteristiche basandosi sulle relazioni tra tutte le variabili. L’analisi univariata considera invece una sola variabile alla volta, producendo risultati ponderati in modo diverso.
L’obiettivo dell’analisi statistica è la categorizzazione e la previsione delle proprietà dei campioni attraverso la generazione di modelli che catturano le informazioni contenute nelle matrici di dati. Nella spettrometria di massa, il rapporto m/z e l’intensità del segnale sono le due variabili più importanti. Nell’NMR selezioniamo i segnali integrati di interesse per l’analisi dei dati.
Nell’ambito dell’analisi multivariata, distinguiamo metodi supervised e unsupervised come i due principali approcci di analisi statistica e machine learning utilizzati per analizzare i dati.
Metodi Unsupervised
I metodi unsupervised non richiedono etichette o variabili di output predefinite. L’obiettivo è esplorare la struttura intrinseca dei dati, trovando pattern, gruppi o caratteristiche rilevanti.
Caratteristiche principali:
- No etichette: Non esistono etichette a priori; i metodi cercano di individuare somiglianze o differenze nei dati.
- Scoperta di pattern: Sono utilizzati per esplorare i dati e identificare raggruppamenti naturali o ridurre la dimensionalità.
- Esempi di metodi unsupervised:
- Clustering: Algoritmi come Hierarchical Clustering dividono i dati in gruppi basati su similarità.
- Riduzione della dimensionalità: Metodi come PCA (Principal Component Analysis) semplificano la rappresentazione dei dati preservandone le caratteristiche principali.
Metodi Supervised
I metodi supervised richiedono un set di dati etichettati, in cui ogni campione è associato a un’etichetta o a una variabile di output nota (ad esempio, classe, valore numerico, ecc.). L’obiettivo principale è costruire un modello che sia in grado di prevedere accuratamente l’etichetta per nuovi dati non etichettati.
Caratteristiche principali:
- Training su dati etichettati: Si utilizza un insieme di dati noti (training set) per apprendere la relazione tra le variabili indipendenti (input) e le etichette (output).
- Predizione: Una volta addestrato, il modello può essere utilizzato per prevedere le etichette di nuovi campioni.
- Esempi di metodi supervised in metabolomica: PLS-DA (Partial Least Square – DIscriminant Analysis)
Analisi delle Componenti Principali (PCA)
L’Analisi delle Componenti Principali (PCA, Principal Component Analysis) è una tecnica di riduzione della dimensionalità utilizzata per semplificare set di dati complessi preservandone le caratteristiche più rilevanti. È ampiamente impiegata in numerosi campi, come la metabolomica, la genetica, l’analisi di immagini, e altre discipline che gestiscono dati ad alta dimensionalità.
Obiettivo della PCA
La PCA mira a trasformare un set di dati originale (caratterizzato da molte variabili correlate) in un nuovo sistema di coordinate, chiamato componenti principali. Queste componenti:
- Sono ortogonali (non correlate tra loro).
- Catturano la varianza massima: la prima componente principale (PC1) cattura la maggior parte della varianza nei dati, seguita dalla seconda (PC2), e così via.
Questo processo consente di rappresentare i dati con un numero inferiore di variabili, riducendo la complessità ma mantenendo la maggior parte dell’informazione.
Come vengono calcolate le componenti principali? Vedi il materiale presente al link “DATA ANALYSIS” sotto per io dettagli. Fondamentalmente, si scelgono le componenti principali che spiegano una frazione significativa della varianza totale (ad esempio, il 95%) e i dati originali vengono trasformati nel nuovo sistema di coordinate formato dalle componenti principali.
Grafici principali nella PCA
1. Score Plot
Lo score plot mostra la proiezione dei campioni nello spazio delle componenti principali selezionate (ad esempio, PC1 e PC2).
- Cosa rappresenta:
- Ogni punto nel grafico corrisponde a un campione nel dataset.
- Le coordinate di un campione sul grafico rappresentano i suoi punteggi (scores), ovvero i valori proiettati sulle componenti principali.
- Utilità:
- Identifica pattern nei dati: ad esempio, raggruppamenti naturali, tendenze, o separazioni tra gruppi (es. malati vs sani).
- Aiuta a rilevare outlier o campioni anomali.
- Esempio:
- Un gruppo di campioni che si aggregano in una regione dello score plot può indicare una similarità tra di essi (ad esempio, appartenenza a una stessa classe).
2. Loading Plot
Il loading plot rappresenta i carichi (loadings), ovvero i coefficienti che descrivono il contributo delle variabili originali (metaboliti, geni, ecc.) alle componenti principali.
In uno studio metabolomico, un loading plot potrebbe evidenziare quali metaboliti sono responsabili della separazione tra gruppi di campioni osservata nello score plot.
Cosa rappresenta:
Ogni punto nel grafico corrisponde a una variabile originale. La posizione di una variabile indica quanto questa contribuisce a ciascuna componente principale.
Un loading plot sotto forma di istogramma è una rappresentazione alternativa dei carichi (loadings) che descrivono il contributo delle variabili originali (ad esempio, metaboliti, geni, segnali) a una specifica componente principale (PC). Invece di visualizzare i carichi come punti o vettori su un piano cartesiano (come accade nel loading plot tradizionale), in un istogramma ogni barra rappresenta il peso di una variabile su una determinata componente principale.
Caratteristiche del Loading Plot a Istogramma
- Asse X:
- Rappresenta le variabili originali (es. nomi o numeri che identificano i metaboliti, geni, o segnali di interesse).
- Ogni barra corrisponde a una variabile.
- Asse Y:
- Indica il valore del carico (loading), che può essere positivo o negativo.
- I valori positivi mostrano una correlazione diretta con la componente principale, mentre i valori negativi indicano una correlazione inversa.
- Altezza delle barre:
- La lunghezza di ciascuna barra rappresenta il contributo della variabile alla specifica componente principale.
- Variabili con barre più alte (in valore assoluto) hanno un’influenza maggiore sulla componente.
Utilità:
Identifica le variabili che influenzano maggiormente le componenti principali. Aiuta a interpretare la biologia o il fenomeno dietro ai pattern osservati nello score plot. Rileva correlazioni tra variabili: variabili vicine o nella stessa direzione possono essere correlate, mentre quelle opposte possono essere anti-correlate.
Qui potete scaricare il materiale utile allo studio dell’analisi dei dati di metabolomica: DATA ANALYSIS
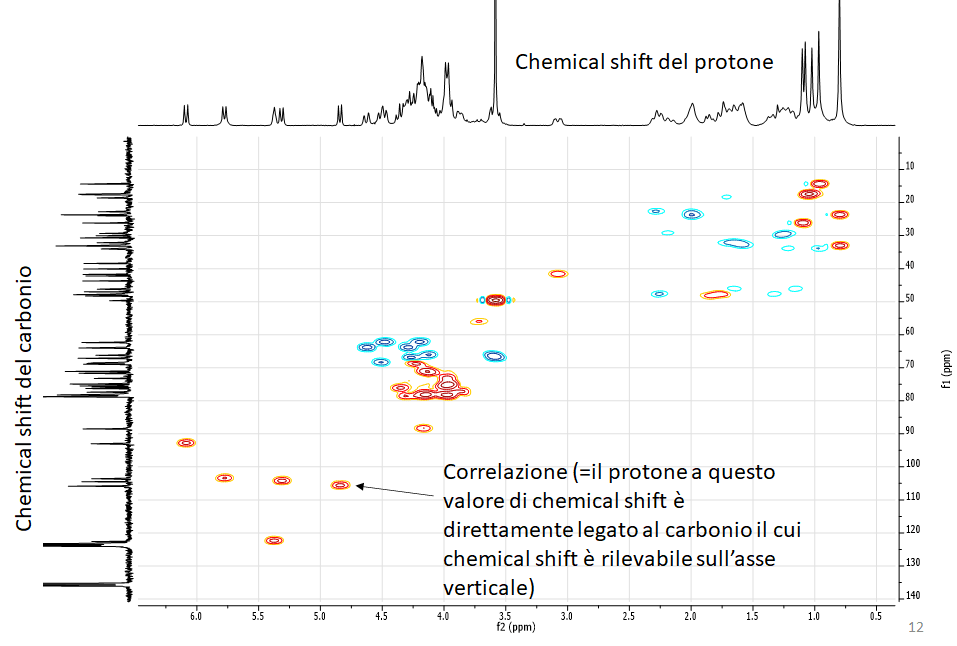
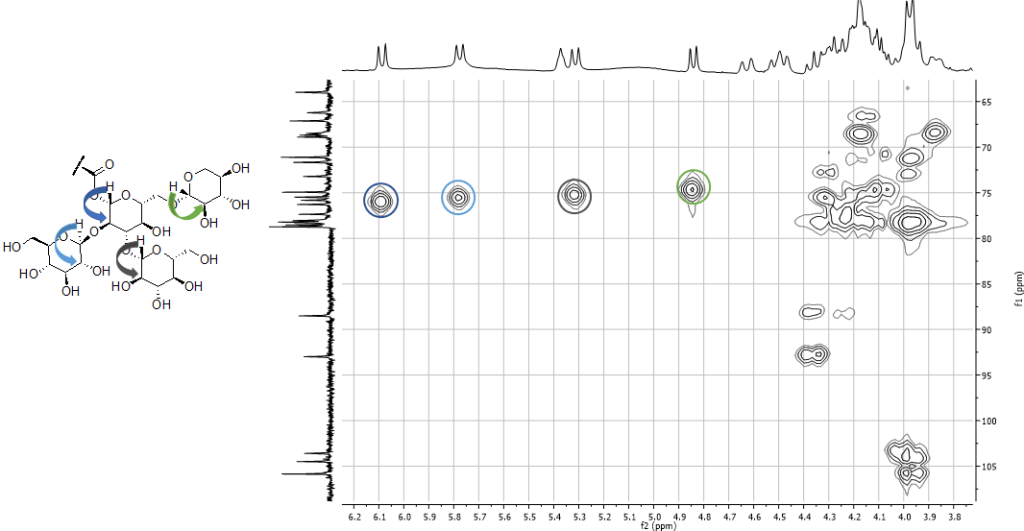
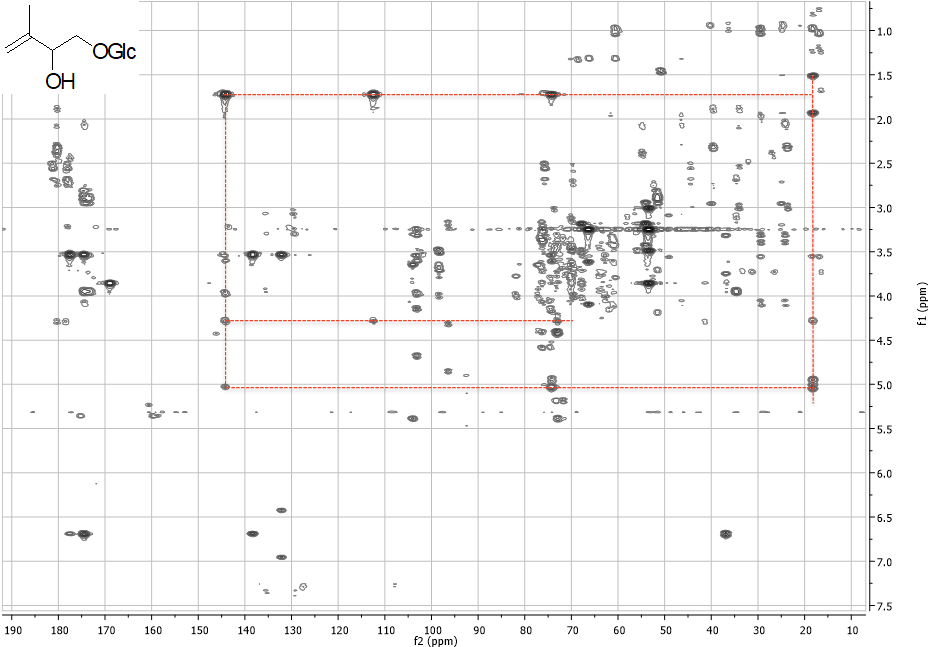
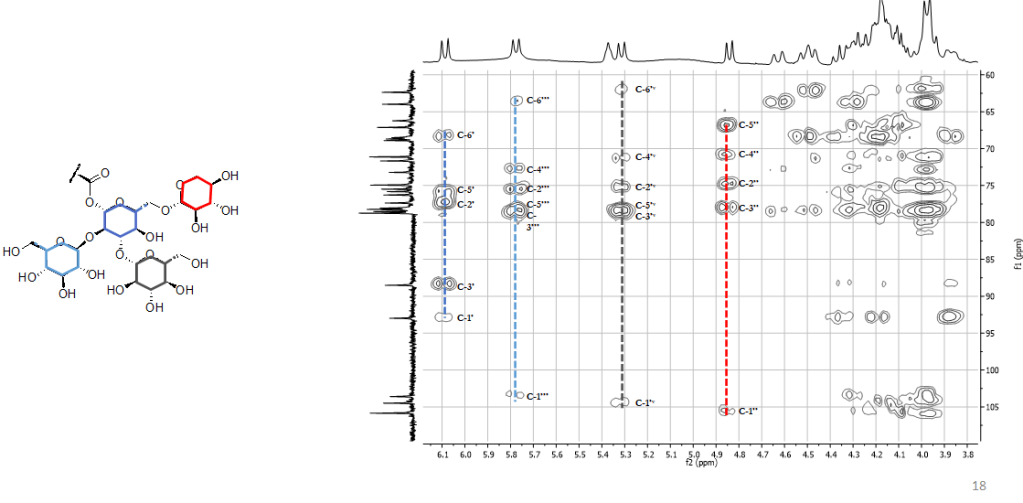
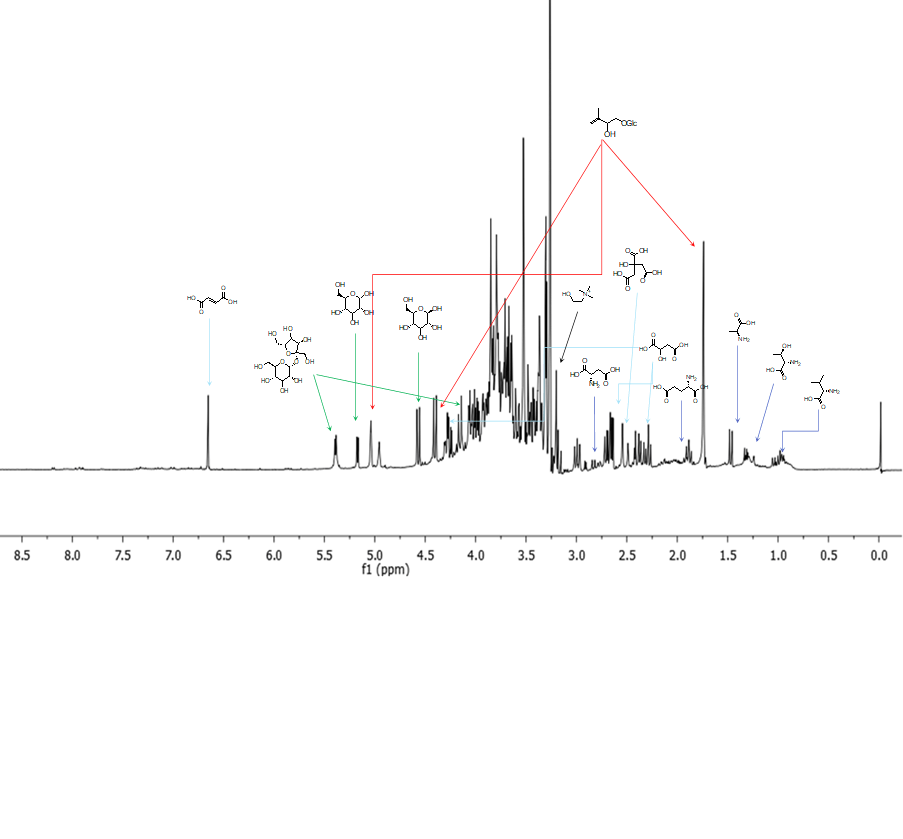
Una volta ottenute le variabili significative, dovremo capire quali metaboliti si “nascondono” dietro queste variabili, ad esempio, analizzando i dati NMR.
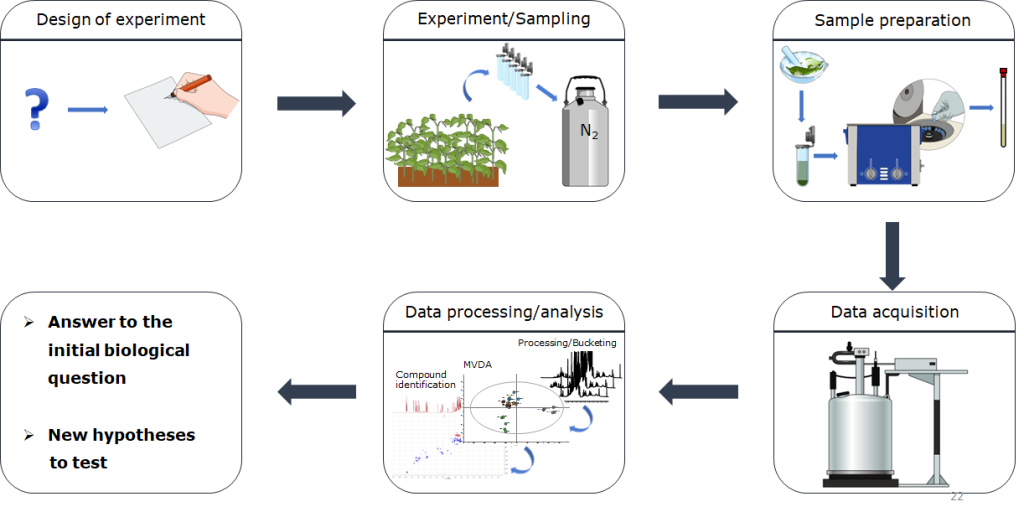
NB: L’analisi metabolomica prevede vari step e ognuno di questi porta con se una certa variabilità.
La standardizzazione delle procedure analitiche, di analisi e presentazione dei dati è di fondamentale importanza per poter “parlare” un linguaggio comune che permetta di confrontare i risultati di esperimenti diversi.
Sul sito della Metabolomics Society, alla pagina dedicata, è possibile leggere informazioni importanti su questo punto. La pagina riporta anche una serie di link a progetti e pubblicazioni.
Oltre a quelle riportate sul sito, vi consiglio anche i “Proposed minimum reporting standards for chemical analysis” e “for data analysis” e l’articolo “Metabolite identification: are you sure? And how do your peers gauge your confidence?”

![]() Ad inizio ottobre si terrà un incontro preliminare di presentazione del CORSO DI METABOLOMICA (insegnamento a scelta – CdLM in Biologia). La data, l’orario e il luogo di questo incontro saranno comunicati successivamente.
Ad inizio ottobre si terrà un incontro preliminare di presentazione del CORSO DI METABOLOMICA (insegnamento a scelta – CdLM in Biologia). La data, l’orario e il luogo di questo incontro saranno comunicati successivamente. ![]() Ulteriori informazioni sono disponibili ai seguenti link: Syllabus
Ulteriori informazioni sono disponibili ai seguenti link: Syllabus

















You must be logged in to post a comment.