Tecniche Analitiche in Metabolomica: NMR

La Spettroscopia di Risonanza Magnetica Nucleare (NMR) è una tecnica che non ha rivali nell’ambito della caratterizzazione strutturale. Questa tecnica spettroscopica, che sfrutta le proprietà magnetiche dei nuclei di alcuni atomi, ci permette di avere informazioni fondamentali sui composti organici. Non tutti i nuclei sono attivi all’NMR, ma tra quelli attivi si annoverano il protone e l’isotopo 13C del carbonio ed è proprio grazie all’applicazione a questi nuclei che otteniamo informazioni strutturali fondamentali per poter identificare la struttura dei composti.
Da uno spettro protonico possiamo dedurre numerose informazioni: il chemical shift ci dà informazioni su quello che è l’intorno chimico, la molteplicità ci dà informazioni sul numero di protoni legati ai carboni vicini (con le costanti di accoppiamento che ci forniscono altre importanti informazioni strutturali), infine l’integrazione ci permette di avere informazioni di tipo quantitativo.
L’utilizzo di techinche 2D-NMR, inoltre, ci dà la possibilità di ricostruire l’intero scheletro delle molecole.
Tutte queste potenzialità sono estremamente utili in metabolomica. Abbiamo visto (e vedremo) in che modo possiamo applicare questa tecnica all’analisi metabolomica.
Intanto, cerchiamo di capire meglio il principio di base su cui poggia questa potentissima tecnicha.
Di seguito, due video che possono aiutarci. Il primo, prodotto dalla Bruker, il secondo da Sciencesketch.
Curiosi anche di sapere come è fatto lo strumento all’interno? Date un’occhiata qui!
Al link seguente è possibile scaricare il materiale di supporto allo studio:
Testi utili per ulteriori approfondimenti:
-D’Ischia “La chimica organica in laboratorio” Ed. Piccin
-Hesse “Metodi Spettroscopici in Chimica Organica”
(entrambi disponibili in biblioteca; in caso di indisponibilità, rivolgersi alla docente)
NMR-based metabolomics
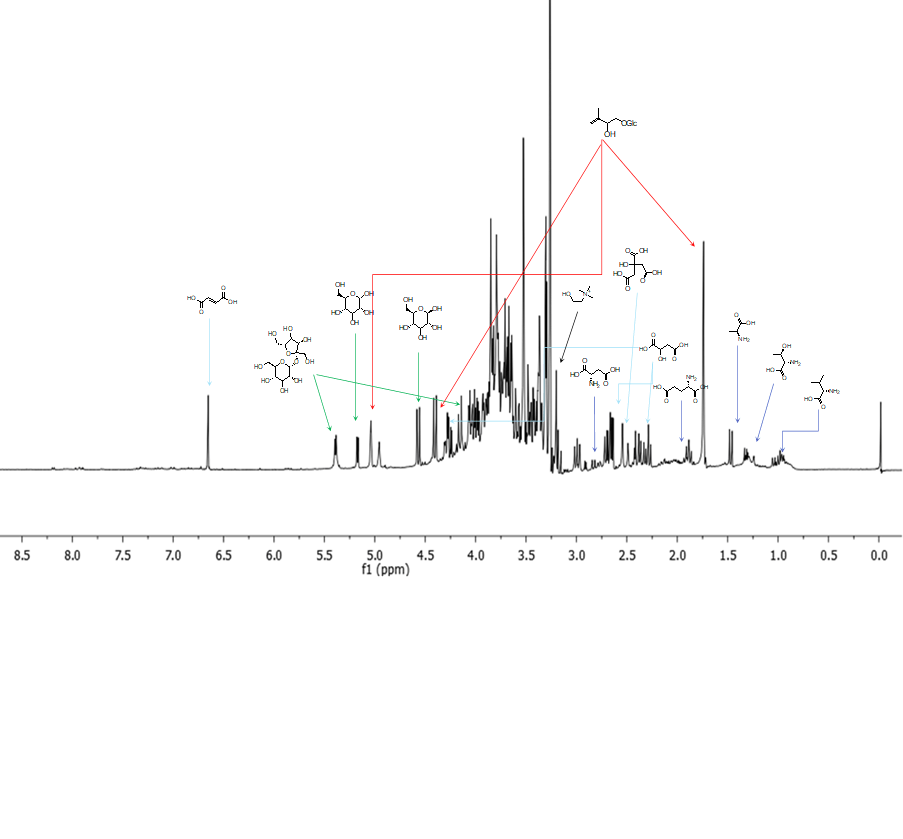
Dall’analisi NMR di un campione si possono dedurre tante informazioni diverse. Ma come si effettua un’analisi metabolomica mediante NMR?
A partire dal materiale liofilizzato, si ottengono gli estratti con una procedura molto semplice che prevede l’estrazione diretta in solventi deuterati (necessari per l’analisi NMR). Questi estratti vengono analizzati, ottenendo gli spettri, che saranno poi processati: questo processing prevede l’apodizzazione, la fasatura, la calibrazione rispetto allo standard intetno e la correzione della linea di base

A questo punto si procede con l’integrazione. Questa è effettuata attraverso il processo di bucketing o binnig: lo spettro si divide in tanti segmenti di lunghezza definita (in genere 0.02 o 0.04 ppm) e si procede ad integrare l’area sotto la curva di ogni bucket. Si ottiene in questo modo una matrice di dati in cui le osservazioni sono i singoli campioni analizzati e le variabili sono i vari bucket, che assumeranno quindi il valore dell’area per quella parte dello spettro in ciascun campione (NB: le aree sono in genere normalizzate rispetto allo standard interno a alla total intensity).

La matrice di dati così ottenuta è sottoposta ad analisi statistica multivariata. Questa sarà utile ad estrarre le informazioni significative dal nostro set di dati.
Una volta identificati i segnali NMR significativi per la nostra analisi, è necessario “tradurre” questi segnali in metaboliti. Si opera a questo punto per step successivi.
Il primo passaggio è quello del confronto con la letteratura e con i database. A questo proposito, è necessario sottolineare come l’NMR sia una metodica altamente riproducibile. In ogni caso, se questa ricerca non ci dà la risposta sperata, si può optare per l’analisi NMR bidimensionale (2D NMR).I metodi 2D NMR più utilizzati in metabolomica sono brevemente descritti di seguito.
COSY (COrrelation SpecroscopY)
Esperimento 2D omocorrelato. Permette di rilevare correlazioni omonucleari 1H-1H tra protoni vicinali e geminali

TOCSY (TOtal Correlation SpecroscopY)
Esperimento 2D omocorrelato. Permette di rilevare sistemi di spin (il trasferimento di magnetizzazione è interrotto da carboni quaternari).

HSQC (Heteronuclear Single Quantum Coherence)
Esperimento 2D eterocorrelato. Permette di rilevare le correlazioni dirette protone-carbonio. Permette quindi di attribuire il valore di chemical shift del carbonio per ciascun carbonio protonato presente nell’estratto (o nella molecola, quando lo spettro si riferisce ad un composto puro)
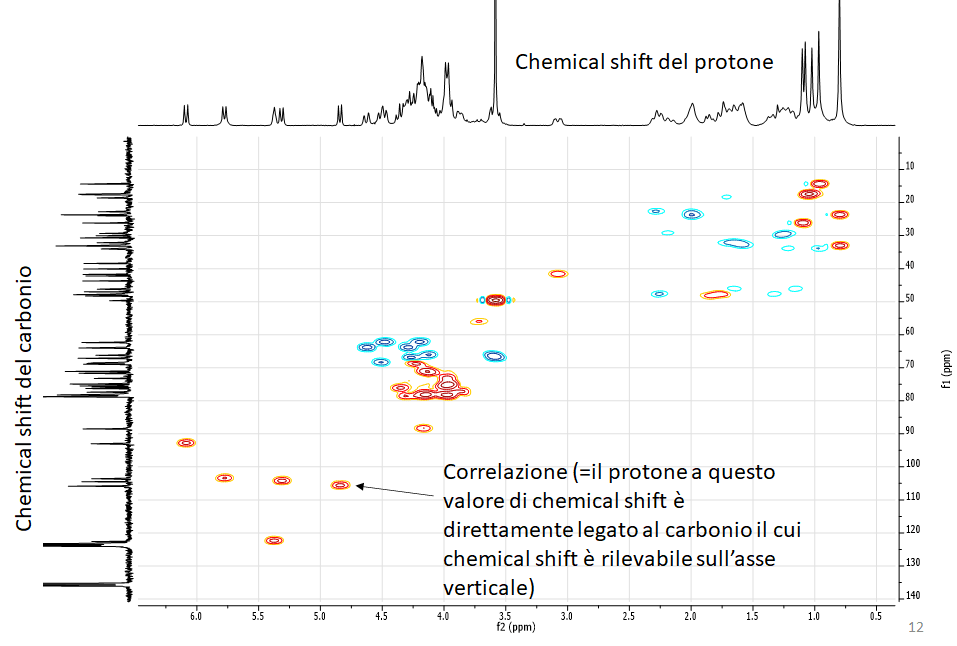
H2BC (Heteronuclear 2 bond correlation)
Esperimento 2D eterocorrelato. Permette di rilevare le correlazioni tra un protone e il carboni vicinale, a patto che quest’ultimo sia protonato.
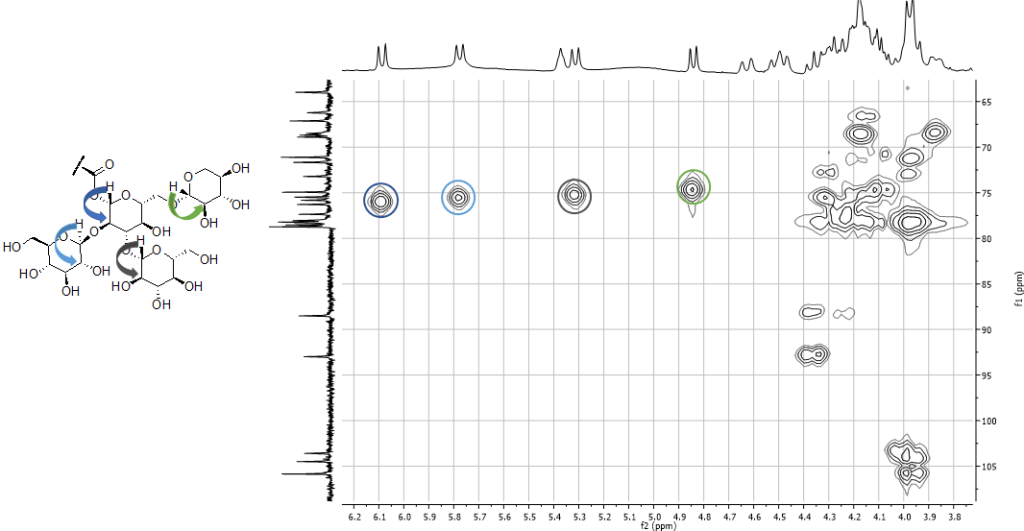
HMBC (Heteronuclear Multiple Bond Coherence)
Esperimento 2D eterocorrelato. Permette di rilevare le correlazioni tra un protone e carboni distanti due, tre o quattro legami. Un esperimento alternativo è noto come CIGAR-HMBC.
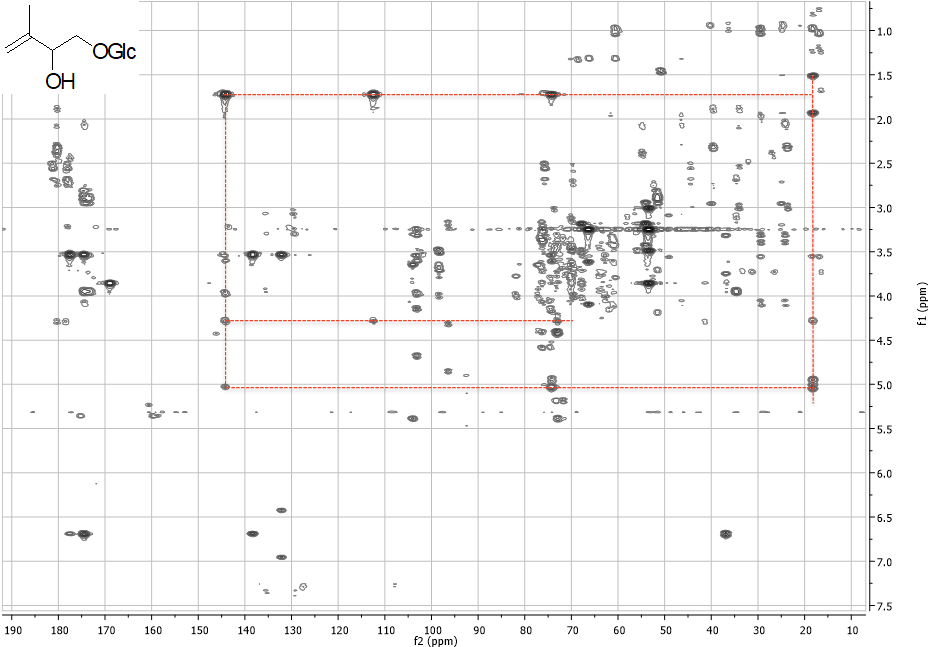
HSQC-TOCSY
Esperimento 2D eterocorrelato. Permette di rilevare sistemi di spin, che in questo caso includono sia i protoni sia i carboni.
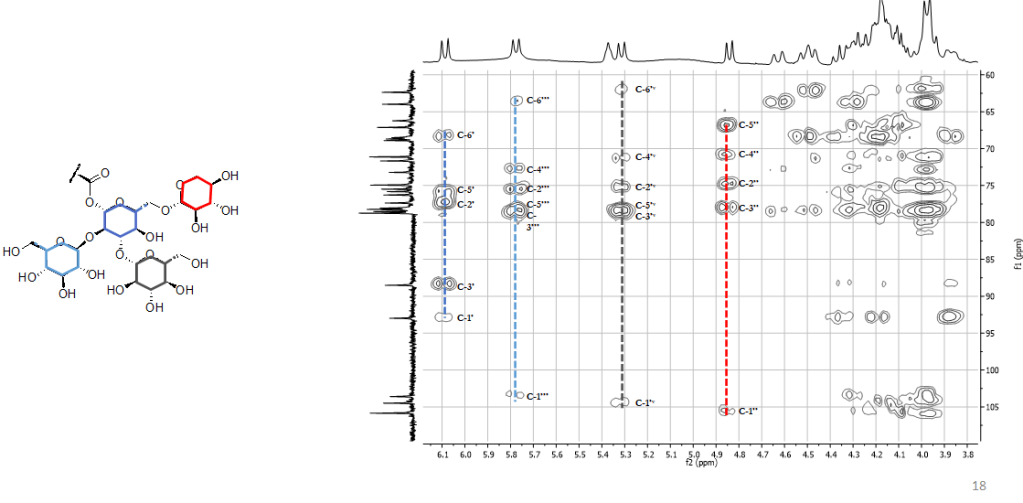
Grazie alla combinazione delle informazioni che si ottengono dai diversi spettri 2D NMR, è possibile identificare i costituenti dell’estratto anche in miscela. Per metaboliti già noti, a questo punto sarà possibile confrontare i dati NMR con quelli riportati in letteratura o con quelli degli standard (a patto che siano acquisiti nello stesso solvente). Per i composti identificati per la prima volta, al fine di confermare la struttura, saranno necessari l’isolamento (che a questo punto sarà facilitato dalle informazioni preliminari in nostro possesso circa la struttura del composto) e la completa caratterizzazione strutturale mediante tecniche spettroscopiche.
Va infine ricordato che oltre ad identificare i componenti dell’estratto, è anche possibile quantificarli dato che l’ 1H NMR è una tecnica quantitativa (se gli spettri sono acquisiti con determinati parametri) e che è sufficiente in questo caso utilizzare uno standard interno a concentrazione nota.

You must be logged in to post a comment.